NSDT工具推荐: Three.js AI纹理开发包 - YOLO合成数据生成器 - GLTF/GLB在线编辑 - 3D模型格式在线转换 - 可编程3D场景编辑器 - REVIT导出3D模型插件 - 3D模型语义搜索引擎 - AI模型在线查看 - Three.js虚拟轴心开发包 - 3D模型在线减面 - STL模型在线切割 - 3D道路快速建模
我在伯贝克学院完成学业后,开始在伦敦国王学院 LIDo 攻读博士学位。我正在使用机器学习研究抗体环(antibody loops)的形状,你可以在 zenodo 上看到所有这些的代码。大部分时间我都在使用 Tensorflow。它为我当前的项目打下了良好的基础,该项目涉及完全不同的东西——显微镜图像。
1、光的问题
简而言之,我们希望能够拍摄细胞和蛋白质的照片,并对它们做更多的事情。由于蛋白质和细胞非常小,我们遇到了光波长和我们可以分辨的物体大小的问题。你可以用电子显微镜做很多事情,但最终你会杀死你看到的一切。进入超分辨率显微镜!
超分辨率使用了许多技巧来绕过光的衍射极限。一个叫恩斯特·阿贝的家伙提出了阿贝极限,它给出了特定波长的最小可分辨距离:
d=λ2nsinθ=λ2NA其中 (NA) 是数值孔径(大约 1.4 或 1.6)。最终,你能做的只有这么多用光来解析,而我们想要观察的很多东西都比这个极限小得多。所以我们需要一些技巧!
维基百科上有很多技术,但在我看来,STORM 和 PALM 是最常用的技术。这个想法是,随着时间的推移,你会拍摄许多图像,然后将它们组合起来,从而创建一个更详细的图像。
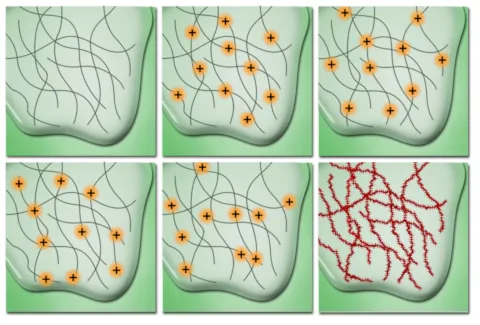
这是可能的,因为蛋白质通常被成像。如果你对一个特定的分子感兴趣,它往往会被标记上一种小抗体或其他化学物质,称为荧光团。当受到另一种波长的激发时,它们会发出特定波长的光。然而,当我们有很多这样的分子时,它们往往会随机发光(或者如果我们想得更具体一点,可以随机发光!)。它们的光也往往有一种模式,可以用一种叫做点扩展函数的东西来建模。考虑到这些,我们可以开始创建图像了。
首先,我们可以拍摄单个点的模糊图像,然后使用点扩展函数找出实际点的位置。想象一下,取一个像素的反向操作,然后在所有方向上将其模糊,例如 10 个像素左右。我们可以很好地猜测原始点的位置和大小。
其次,由于荧光团仅随机发光,因此遮挡问题会减少。想象一下,两个非常接近的荧光团同时发光。它们会模糊成一个,我们很难将它们分开。当然,如果一个荧光团先于另一个荧光团闪烁,我们会获得更多信息来帮助我们解决每个问题。
这种技术的问题在于我们需要拍摄多张图像,而不仅仅是一张。我们需要尽可能快地使用点扩展函数和我们可能使用的任何其他计算机视觉过程。这就是机器学习和工程的用武之地。
我们经常使用这些图像来了解细胞处于什么状态或具有什么形状。如果我们能够在特定的分子上绘制点,我们就能了解它的结构,这在生物学中是非常方便的。
2、让我们把问题简单化
现在,让我们把事情简单化。如果我们知道我们在寻找什么类型的东西,那么在图像中发现它会更容易。一种这样的简单蛋白质被称为 CEP-152,它看起来像一个圆环。所以现在让我们假设我们正在寻找圆环状的东西。如果我们假设我们有一个圆环状蛋白质的图像,我们能知道它的方向吗?
事实证明我们可以!
3、进入 PyTorch
PyTorch 是原始 Torch 库的演变,用于处理繁重的数学和机器学习。它与 Tensorflow 略有不同,我想稍微改变一下。我非常喜欢它,而且它相当容易上手。
计划是拍摄一张图片,将其通过卷积网络并创建另一张图片,然后比较这两张图片。这应该会导致一个错误,我们可以将其向后传递到网络中以更新权重,并希望了解有关图像的一些信息。
我们将问题分为两部分。第一个是卷积网络,它拍摄一张图片并产生 3 个值:x 旋转、y 旋转和 z 旋转。这部分相当简单,可以在 PyTorch 网站上找到这类事情的几个例子。
4、可微分渲染器
问题的后半部分更加棘手,需要我们获取一组点和三个旋转并创建 2D 图像。从哪里开始呢?
值得庆幸的是,我记得我使用 pxljs 时的所有事情,并且 3D 图形的渲染管道有很好的文档记录。获取 3D 点(实际上是 4D,因为它们有 x、y、z 和 w 值),将它们乘以大约 4 个不同的矩阵,最终将得到需要打开的图像中像素的 2D 坐标。
这组矩阵乘法是可微分的,因此可以在 PyTorch 中实现并用于损失函数。你可能认为一切都已排序?实际上,不是。
如果你像我一样是一名程序员,你会获取漂亮的 2D x、y 像素位置并将这些值用作图像的索引。例如,获取 y,将其乘以图像的宽度并添加 x,然后你就得到了一个索引。将其设置为 1,然后砰!完成。好吧,这是不可微的。真讨厌!
但有办法!我们可以将二维向量 (x,y) 拆分成 2 个二维矩阵,大小与我们的输出图像相同。第一个矩阵只包含 x 值,第二个矩阵只包含 y 值。你会问这有什么用?
好吧,如果我们从一个特殊的索引矩阵中减去每个矩阵并将这两个矩阵合并,我们会得到一个偏移矩阵,其中我们感兴趣的点设置为 0,而所有其他点都设置为与该点的距离。有了这些信息,我们就可以创建最终图像。事实证明,索引矩阵在这种排序问题空间中非常常见。以下是 Python 中的代码:
# Create our index matrix for x and y.
numbers = list(range(0,size[0]))
square = [ numbers for x in numbers]
xs = torch.tensor(square, dtype = torch.float32)
ys = xs.permute([1,0])
# Now expand out our x and y value
ex = px.expand(points.shape[0], xs.shape[0], xs.shape[1])
ey = py.expand(points.shape[0], ys.shape[0], ys.shape[1])
offset_x = xs - es
offset_y = ys - ey
final_matrix = torch.abs(offset_x) + torch.abs(offset_y)如果这还不够清楚,下面是我们要用一个非常小且简单的 3 x 3 图像尝试做的事情。假设我们要将像素 (1,2) 设置为白色:
index x matrix x matrix final x matrix
0 1 2 1 1 1 -1 0 1
0 1 2 - 1 1 1 = -1 0 1
0 1 2 1 1 1 -1 0 1
index y matrix y matrix final y matrix
0 0 0 2 2 2 -2 -2 -2
1 1 1 - 2 2 2 = -1 -1 -1
2 2 2 2 2 2 0 0 0
abs final x abs final y final final
-1 0 1 -2 -2 -2 3 2 3
abs -1 0 1 + abs -1 -1 -1 = 2 1 2
-1 0 1 0 0 0 -1 0 1就这样!一个矩阵,其中我们想要的点设置为 0。我们只需要设置一次索引矩阵。使用 PyTorch 中的函数创建 x 和 y 矩阵相当容易。我得到了一些帮助来解决这个问题。我花了几天时间撞墙。最好的建议?不要这样做。向你的主管和同学寻求帮助。
5、模拟数据
我们可能应该先从一些地面真实数据开始。让我们确保一切都正常。我们有一个很好的圆环模型和一个函数来在顶点处创建漂亮的高斯斑点。让我们生成大量图像,然后将它们传递到我们的网络中,看看它是否可以恢复角度。

让我们从最右边的开始。它的角度分别为 1、-67 和 -77 度。让我们看看最终结果是什么?
Output rotations: 67.82590264705848 -22.634898569774585 -24.638662628844763看起来不太好,但我们不要太急。实际图像是什么样的?

这是怎么回事?嗯,圆环是相当对称的,不是吗?另外,旋转,比如说 -180,与旋转 180 是一样的,所以有很多组合实际上会产生相同的图像。所以最后的结果还不错。
6、下一步计划?
不对称是下一个要测试的东西。这减少了可接受的解决方案的数量。我们假设某种蛋白质呈圆环形状,这是非常有限的,而且是一个相当困难的假设,所以我们最终需要找到一种方法来解决这个问题。
原文链接:Using PyTorch to find the orientation of a torus
BimAnt翻译整理,转载请标明出处





